2019-05-16
历时2年做出的实验结果,终于在文章投出的时候轻松片刻!但是,一个不留神就可能被要求补数据!
审稿人驳回RNAi文章,常常提出这样的建议:

What?RNAi的坑到底在哪?
为了让大家避免“万事具备、只欠东风”的尴尬,今天就给聊一聊RNAi研究怎样避坑。
RNA干扰(RNA interference, RNAi)是指在进化过程中高度保守的、由双链RNA(double-stranded RNA,dsRNA)诱发、同源RNA高效特异性降解的现象,通常用于降低特定基因的RNA水平。
但在实际应用中,众多科研工作者只注重功能变化,忽略了RNAi技术缺点——脱靶(Off-Target:非靶基因抑制),若脱靶基因与靶基因位于同一信号通路或者与表型相关,则容易出现假阳性,对实验结果产生干扰,那么如何在实验设计前端就避免呢?我们先看下靶点设计过程:
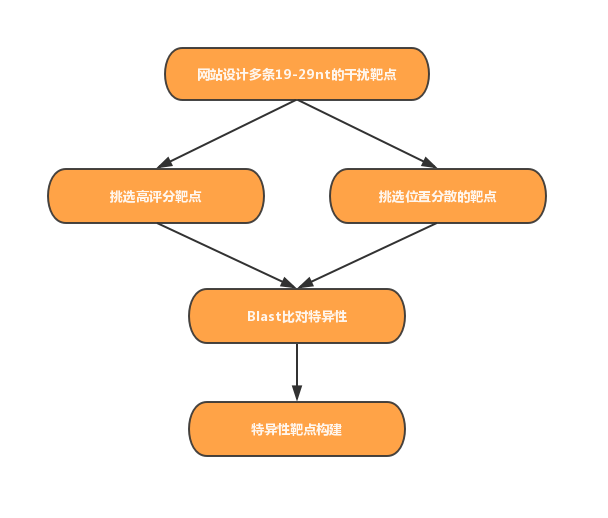
图1
防止脱靶的第一步是Blast比对,即将靶点与对应物种核酸数据库比对,确保其与非靶基因含有至少3个以上碱基错配。
这就够了吗?NO!
按照Nature的标准,一个严格的RNAi介导knockdown实验要有6个对照:
1.转染试剂对照(监控转染及培养条件对结果的影响);
2.nonsense siRNA对照(监控外源核酸本身对结果的影响);
3.positive siRNA对照(监控假阴性);
4.重复对照(也叫off-target 对照,也就是利用至少2个靶点的siRNA同时实验,由于靶点序列不同,脱靶至同个基因的概率几乎为0,所以2个siRNA互为off-target control,当两者的表型相同时,才有可能是特异性的knockdown效应);
重复对照的另一个途径是RNAi搭配crispr/cas9使用,分别在RNA、DNA水平的敲减操作既实现off-target control,又因为使用了不同的技术路线,提高了文章亮点[1]。
5.rescue对照(也称回复突变或者拯救实验:将siRNA与靶基因结合处进行同义突变,并将该序列在已knockdown的细胞内过表达,看是否有性状的逆转,若逆转,表示性状的改变同靶基因相关;若无逆转,则性状的改变是因为脱靶造成的);
6.全表达组扫描对照(就是转录/表达芯片扫描,以最终确定表型不是由于off-target造成)。
以上六组对照,前三组是基础;第6组鲜有文献涉及,可以舍去;4或者5任做一组即可,若提交的文章不含组4、5,审稿人会依据自己的偏好做指定要求。两种方法对应的实验分组如下:
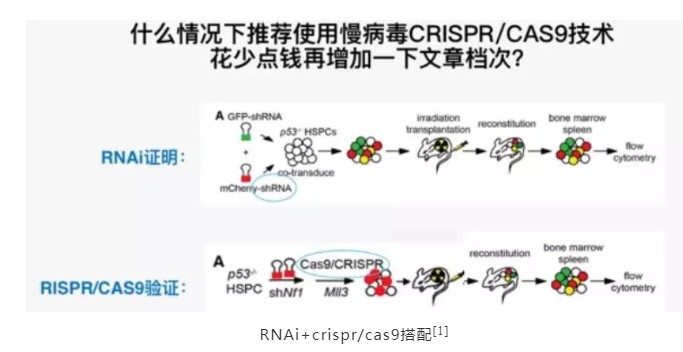
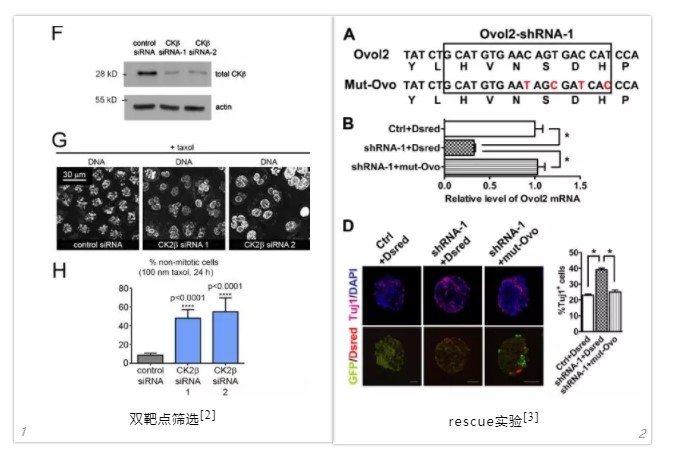
审稿人对脱靶的担忧是多余的吗?我们再看看脱靶数据及原因:
一 、siRNA剂量引发脱靶效应
高浓度的siRNA与非靶基因结合概率增大,从而诱发off-target。
Caffrey[4]做了不同浓度的脱靶研究,以图2红框内前三个STAT3靶点为例,25nM、10nM、1nM的siRNA分别造成38、1、0个脱靶。由此可见,1~2倍的siRNA剂量差异可引发指数级的脱靶效应。而大多数全基因组和生物筛选的siRNA浓度通常为25nM至100nM,远高于文章使用的剂量,且载体表达的siRNA依靠U6、H1启动子表达,siRNA剂量因启动子在不同细胞活性差异以及siRNA本身结构的影响,无法精确控制siRNA浓度。
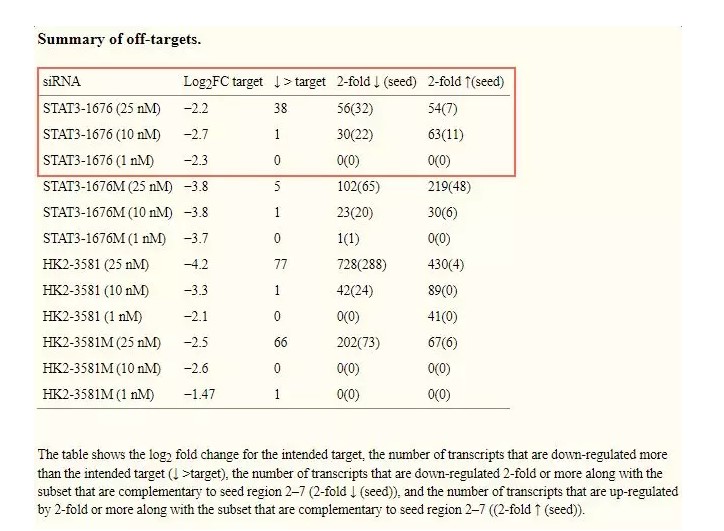
图2
二、siRNA种子区引发脱靶效应
miRNA主要依靠种子区(seed region,指成熟体5’端 2-8个碱基)与靶基因的3'UTR区结合,抑制翻译。siRNA同miRNA产生机制类似(图3),也可通过种子区结合特征产生非靶抑制现象。图2数据显示,非靶基因下调总数/符合种子区特征:56/32,30/22(以前两个为例);Jackson[5]也论述了脱靶效应与seed region的对应关系(图4),seed region结合占脱靶比率超过50%(而脱靶引发的上调不符合种子区结合原则)。
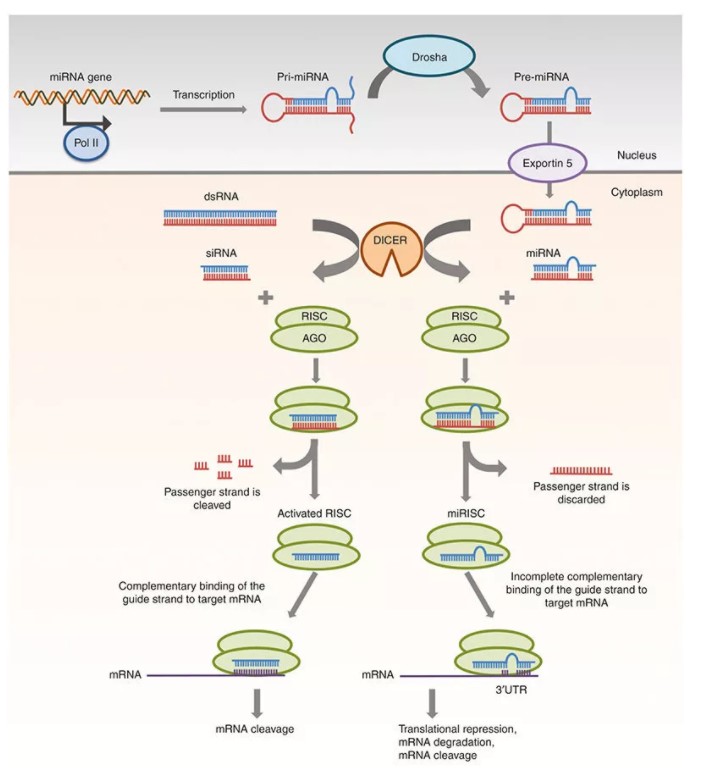
图3
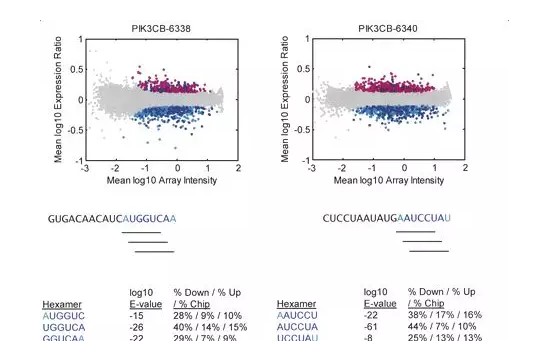
浅蓝色表示下调的转录物
红色表示上调的转录物
灰色表示无显著变化的转录物
深蓝色表示符合种子区结合的下调转录物
序列表示靶点并标记出种子区
图4
RNAi天生带有原罪,载体表达的siRNA剂量不可控;siRNA依靠部分碱基结合非靶基因诱发的脱靶效应无法避免;经过dsRNA剪切产生的siRNA正义链也可参与脱靶效应……因此,实验设计的合理性至关重要。
如果您在做这方面的研究,但是还未做4或5的对照,小编强烈建议您赶紧补充一下吧,以免投稿后再补充数据。
如果您有相关问题,请给我们留言!吉凯病毒专家在您身边哦~
参考文献:
1.MLL3 Is a Haploinsufficient 7q Tumor Suppressor in Acute Myeloid Leukemia
2. The tumor suppressor CDKN3 controls mitosis
3. Ovol2 knockdown phenotype is rescued by expression of shRNA-resistant mutant Ovol2
4. siRNA Off-Target Effects Can Be Reduced at Concentrations That Match Their Individual Potency
5.Widespread siRNA “off-target” transcript silencing mediated by seed region sequence complementarity
© 2023 GENECHEM All RIGHTS RESERVED .
© 2023 GENECHEM All RIGHTS RESERVED .
